劍橋大學「新冠病毒分為ABC株」理論,被基因學家批評「錯漏百出」
劍橋大學彼得.福斯特(Peter Forster)所領導的研究團隊日前(4 月 10 日)於《美國國家科學院院報》(PNAS)發表題為《SARS-CoV-2 基因圖譜之系統發生網絡分析》的研究論文,重塑新冠病毒(SARS-CoV-2)之早期演化路徑,「有助了解新冠病毒傳播起源」。
研究團隊於全球共享流感數據倡議組織(GISAID)下載 160 個完整新冠病毒基因圖譜,使用系統發生網絡分析(phylogenetic network analysis),分類出三種新冠病毒「變種」,研究團隊將其名為 A、B 及 C。A 株最接近野生動物界的病毒宿主(蝙蝠冠狀病毒 RaTG13),被認為最有可能是新冠病毒起源。原始A株主要發現於武漢病人身上,而 A 株的變種則被發現於居於武漢的美國人、美國及澳洲病人;B 株則遍佈東亞;而 C 株主要散播於歐洲區域,亦見於新加坡、香港及南韓。研究團隊認為:「(這項研究)有助辨別新冠病毒(在不同區域)的源頭,可以對之隔離以阻止病毒進一步散播。」
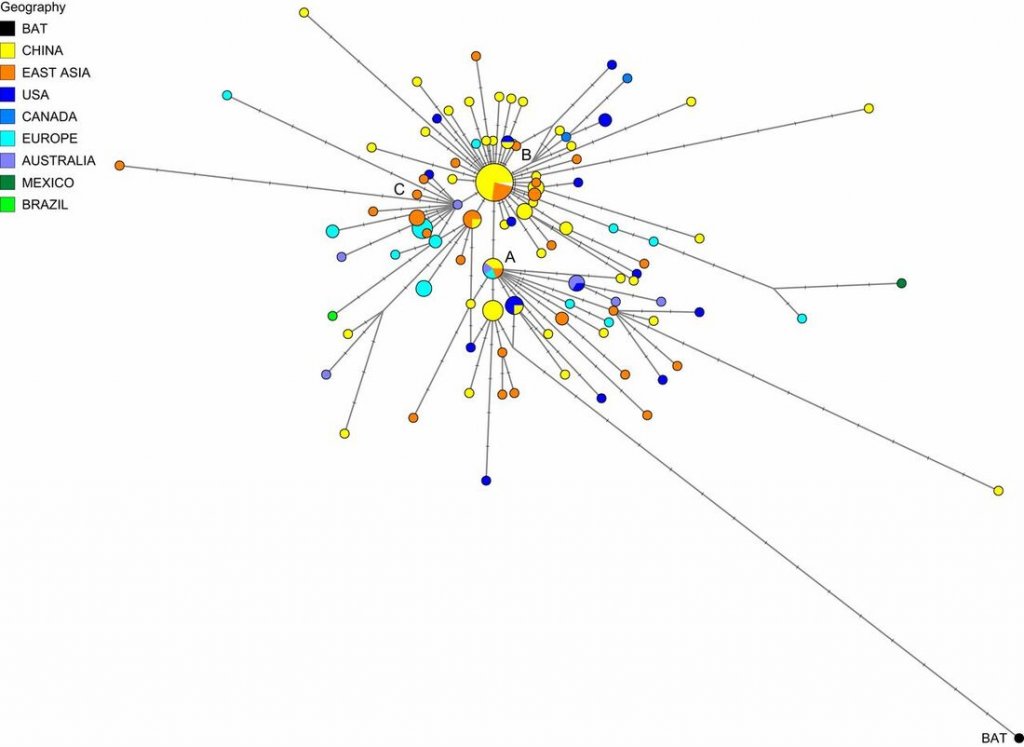
(圖一) 彼得.福斯特團隊以 160 個新冠病毒基因圖譜重構演化關係,A 株原種於武漢病人身上發現、A 株變種於居於武漢的美國人、美國、澳洲病人身上發現、B 株於東亞廣泛傳播、C 株主要於歐洲。
彼得.福斯特團隊的論文引起極大回響,不少媒體甚至錯誤理解論文指出美國有可能為病毒起源,又或者 ABC 三株針對不同人種和群體。隨著時間推進,病毒本來就會因為自然基因變異,演化出不同亞種,但現今科學界仍未發現新冠病毒演化出任何趨勢針對某特定人種。
更重要的是,論文面世僅僅兩日已引起多位流行病學家及基因學家的批評。
原始數據已過時
第一,原始論文只於 3 月 4 日分析了 GISAID 當中的 160 個新冠病毒基因圖譜,並未提出論據為何不使用當時已存在的其餘 100 多個基因圖譜。截至今日(4 月 11 日),GISAID 已發表了超過 3,500 個基因圖譜。GISAID 是一個學術界共享數據的平台,科學家透過共同分析數據並發表文章,互相尊重學者對數據的貢獻。這份論文已經不符合現時新冠病毒的最新發展。
不恰當的假設
第二,原始論文使用了不合適的研究方法和假設,得出無效的結論。GISAID 科學顧問委員會成員、愛丁堡大學分子演化學教授安德魯.蘭博(Andrew Rambaut)批評:「(彼得.福斯特團隊)使用蝙蝠冠狀病毒RaTG13作為外群(outgroup)來判斷新冠病毒起源是錯誤的。」
科學界判斷一組物種的起源有幾種方法,其一是使用外群來定根(outgroup-based rooting)。以圖二為例,內群是一組相似的物種,隨時間演進衍生出物種A 至 D,而物種 A 最接近內群的根(共同祖先)。以新冠病毒情況,如果外群與一組 A 區的病毒株相鄰,則可被判斷為 A 區最有可能為病毒起源。
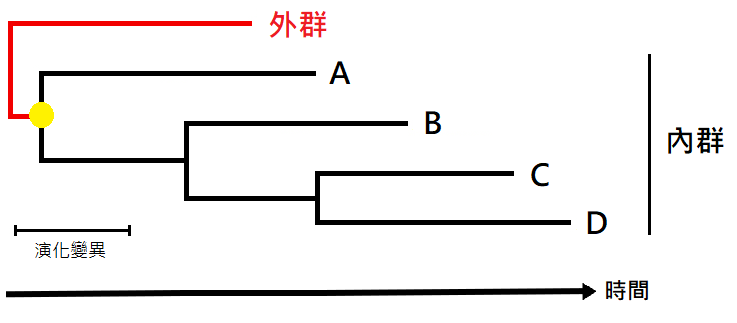
(圖二) 「外群定根法」例子,內群中的 A 最接近外群和內群的根(黃點),因此最有可能是內群中最原始的分支。
安德魯.蘭博以原始論文所使用的外群(蝙蝠冠狀病毒 RaTG13),模擬出外群與所有已知新冠病毒的演化關係(圖三)。

(圖三) 安德魯.蘭博以彼得.福斯特團隊所使用的外群(蝙蝠冠狀病毒RaTG13),以最大似然估計(maximum likelihood) JC69 模型,模擬新冠病毒與外群的演化關係,紅點為外群。
由圖三可見,蝙蝠冠狀病毒 RaTG13 與其餘所有新冠病毒距離極遠,衍生出長枝吸引效應(long branch attraction),在此效應下,即使兩枝病毒株極不相似,仍會被錯誤歸類相近,而由於所有新冠病毒株互相更為接近,「RaTG13 可被置於幾乎任何新冠病毒的鄰枝」,亦即任何地方的新冠病毒株均有可能被錯誤當作起源。因此,研究團員並不應使用 RaTG13 作外群定根,而應考慮利用分子時鐘模型及其他假設來判斷病毒起源。
論文發表過程成疑
第三,論文的發表過程被質疑。論文其中一位共同作者為考古學家科林·倫福儒(Colin Renfrew),他亦為美國國家科學院的外籍成員。根據《院報》論文發表指引,學院成員有權每年兩次「貢獻提呈」(contributed submission)來發表自己的論文,自行選擇論文評審。兩位評審 Toomas Kivisild 以及 Carol Stocking 均並非流行病基因學家,引起其他學者質疑其評審能力。
「錯漏百出,令人沮喪」
安德魯.蘭博於其推特批評:「(彼得.福斯特團隊的論文)錯漏百出,令人沮喪。他們使用其他學者未公開發表的研究數據(GISAID),以及錯誤的研究假設得出錯誤的結論,竟然被頂尖期刊所接受……(他們)無視了大批學者於基因圖譜上及起源分析上的努力及貢獻。」多位新冠病毒基因專家對此表示同意,並批評《院報》的審稿水準,當中包括英國伯明翰大學微生物基因及生物信息學教授尼克.勒曼(Nick Loman)。
新冠病毒的起源及傳播
Nextstrain(https://nextstrain.org)是一個建基於 GISAID 的實時病毒演化監察平台,每星期發表研究報告更新現時科學界對新冠病毒的認知,根據由特雷弗.貝德福德領導於 4 月 10 日發表的最新報告,分析 3,160 個基因圖譜,加上分子時鐘(molecular clock),結論「所有流行中的新冠病毒分株的共同祖先最有可能出現於十一月下旬至十二月上旬的中國武漢。這項發現與中國與亞洲新冠病毒大流行第一個月的情況吻合。」
隨著新冠病毒於亞洲爆發,病毒由一至二月開始散播至北美、歐洲及大洋洲,但並未引起大流行及注意,二月至三月於北美及歐洲的病毒開始大量傳播,並於全球大流行,於最近開始傳播回亞洲。
科學家強烈呼籲大眾要小心應對新冠病毒大流行,應時刻保持社交距離及良好個人衛生,如無必要外出應留在家中,減慢病毒於社區傳播。
資料來源:
- Forster, P., Forster, L., Renfrew, C. & Forster. M. (2020) Phylogenetic network analysis of SARS-CoV-2 genomes. PNAS, 202004999 (early).
- University of Cambridge. COVID-19: genetic network analysis provides ‘snapshot’ of pandemic origins.
- Rambaut, A. “There are many things that are terribly wrong about this paper. Both in its content, findings and route to publication.” Twitter
- Bedford, T. et al. (2020) Genomic analysis of COVID-19 spread. Situation report 2020-04-10. Nextstrain.
- Tang, X. et al. (2020) On the origin and continuing evolution of SARS-CoV-2. National Science Review, nwaa036.
- Hadfield et al. (2018) Nextstrain: real-time tracking of pathogen evolution. Bioinformatics.